Identificare i casi di diabete tipo 2 in una rete di fonti di dati eterogenee: la strategia del Progetto European medical information framework (Emif)
Giuseppe Roberto e Rosa Gini per conto di EMIF-Platform Consortium
Agenzia Regionale di Sanità della Toscana, Osservatorio di Epidemiologia, Firenze
SUMMARY (Identifying cases of type 2 diabetes in an heterogeneous data source network: strategy from the EMIF project) - As a proof-of-concept of a novel standard data derivation procedure for the execution of multi-national, multi-data source studies, type 2 diabetes cases were identified in a set of heterogeneous data sources from 4 different European countries. Standard algorithms (components), each based on one single data domain among diagnoses, drugs, utilization of diagnostic tests and laboratory results, were generated and extracted. Components were then used as building blocks to create data-source tailored case-finding algorithms. This strategy facilitated transparent documentation, benchmarking of data sources and interpretation of study findings.
Key words: database; electronic health records; type 2 diabetes
giuseppe.roberto@ars.toscana.it
Introduzione
L’identificazione di una popolazione d’interesse all’interno di una fonte di dati sanitari informatizzati si fonda sull’applicazione di un algoritmo. Tale algoritmo viene scelto sulla base sia delle caratteristiche della fonte di dati utilizzata (ad esempio, informazioni disponibili) sia del quesito di ricerca a cui lo studio intende rispondere (1, 2). Tuttavia, la scelta dell’algoritmo d’identificazione può avere un impatto significativo sulle caratteristiche della popolazione di soggetti identificata (3, 4) e, pertanto, deve essere tenuta in considerazione per contestualizzare e discutere correttamente i risultati dell’analisi.
Negli studi internazionali basati su reti di fonti di dati eterogenee tra loro, la variabilità degli algoritmi locali d’identificazione aumenta insieme all’eterogeneità delle fonti di dati interessate (5), poiché possono rendersi necessari algoritmi specifici per ciascuna fonte di dati (1). In questo tipo di studi, quindi, un processo trasparente di documentazione e valutazione degli algoritmi locali d’identificazione diviene essenziale per la corretta interpretazione dei risultati ottenuti da ciascuna delle fonti di dati utilizzate (6, 7). All’inizio del 2013 è stato lanciato il progetto European Medical Information Framework (EMIF) al fine di creare una infrastruttura informatica (EMIFPlatform) per il riutilizzo in forma aggregata e scalabile di dati medico-sanitari provenienti dalle fonti già esistenti sul territorio europeo (www.emif.eu/about/emif-platform). Sulla base delle precedenti esperienze europee nel campo degli studi internazionali multidatabase (5, 8), nell’ambito del progetto EMIF è stata ideata una strategia innovativa per la generazione di algoritmi d’identificazione specifici per fonte di dati in forma standardizzata. Questa strategia permette di facilitare l’esecuzione di studi osservazionali di qualità utilizzando in modo combinato fonti di dati con caratteristiche eterogenee tra loro. L’obiettivo di questo articolo è quello di descrivere tale strategia, utilizzando parte dei risultati ottenuti dalla sua prima applicazione pratica (9) in cui, a titolo esemplificativo, sono stati identificati i casi di DM2 nella popolazione adulta.
Materiali e metodi
Fonti di dati - Ai fini di questo articolo sono state considerate 6 fonti di dati che collaborano allo sviluppo della EMIFPlatform. Queste 6 fonti di dati raccolgono informazioni da 4 differenti Stati europei (Italia [I], Olanda [NL], Regno Unito [UK] e Danimarca [DK]) e possono essere classificate in due categorie principali: fonti di dati provenienti dalla medicina generale (MG), ovvero THIN (abbreviazione ai fini dello studio: MGUK), HSD (MG-I) e IPCI (MG-NL), e fonti di dati di tipo amministrativo basate sul record linkage (RL) di differenti flussi, ovvero ARS Toscana (RL-I), PHARMO (RLNL) e AUH (RL-DK). MG e RL differivano l’una dall’altra, anche all’interno delle due categorie, in termini di domini di dati disponibili (ad esempio, diagnosi, prescrizioni farmaceutiche, risultati di laboratorio) e tipologia d’informazioni registrate in ciascun dominio, setting assistenziale in cui i dati erano raccolti (ad esempio, assistenza primaria, secondaria o ospedaliera), terminologie di codifica (ad esempio, per le diagnosi erano utilizzate 4 terminologie differenti: ICD9CM, ICD10, ICPC e READ) e organizzazione del servizio sanitario da cui i dati originavano.
Popolazione e disegno di studio - La popolazione di studio in ognuno dei 6 database corrispondeva a tutti i soggetti attivi al 1° gennaio 2012 (data indice) che a quella data avevano più di 15 anni d’età. È stato effettuato uno studio descrittivo, trasversale, retrospettivo, multidatabase in cui, in ciascuna fonte di dati, sono stati individuati i pazienti con DM2 attraverso l’applicazione di differenti combinazioni logiche di algoritmi d’identificazione. L’applicazione degli algoritmi utilizzava tutto il tempo persona a disposizione per ciascun individuo prima della data indice.
Generazione di una lista di algoritmi componenti - Sulla base di una definizione clinica condivisa di DM2 (10), è stata generata una lista di algoritmi standard, detti algoritmi componenti (Tabella 1), utili all’identificazione dei soggetti con DM2 nelle fonti di dati considerate. Ogni componente si basava sull’utilizzo di record appartenenti a uno solo tra i seguenti domini di dati: diagnosi, utilizzo di farmaci, utilizzo di test diagnostici, risultati di laboratorio; all’interno del dominio delle diagnosi i componenti erano ulteriormente distinti in base al setting assistenziale in cui la diagnosi era formulata (assistenza primaria, secondaria, ospedaliera, altro). La lista di componenti è stata creata attraverso un processo iterativo basato su una strategia di tipo top-down/bottom-up (1). Ciascuna organizzazione partecipante allo studio ha identificato, a livello locale, un esperto riguardo l’identificazione del DM2 nella propria fonte di dati. Sia gli algoritmi reperiti in letteratura, sia gli algoritmi suggeriti dagli esperti locali sono stati scomposti nei diversi componenti. Per l’armonizzazione semantica delle terminologie di codifica è stato utilizzato lo Unified Medical Language System (8).
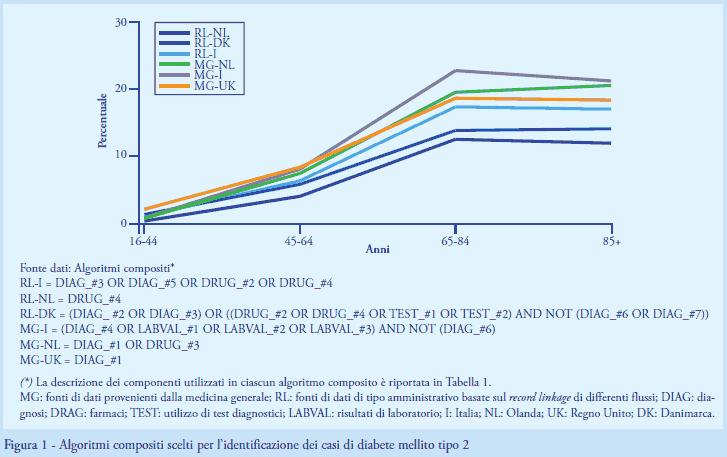
Estrazione e analisi dei dati: la strategia degli algoritmi componenti - Gli esperti locali hanno selezionato ed estratto singolarmente tutti quei componenti considerati utili per l’identificazione del DM2 nella propria fonte di dati. I ricercatori hanno esaminato gli algoritmi componenti estratti sia singolarmente sia in combinazione logica tra loro attraverso l’utilizzo di operatori booleani (OR, AND, AND NOT), ovvero utilizzandoli come criteri di inclusione, di raffinamento o di esclusione ((≥2 prescrizioni di antidiabetici diversi da insulina in 365gg OR ≥2 valori HbA1c >6,5%) AND NOT ≥1 diagnosi DM2). Attraverso questa strategia degli algoritmi componenti, i ricercatori hanno costruito algoritmi d’identificazione più complessi (algoritmi compositi) (Figura 1). Gli esperti locali hanno infine scelto l’algoritmo composito più adatto all’identificazione del DM2 nella propria fonte di dati, sulla base della propria esperienza preesistente e dei suggerimenti dei ricercatori.
Risultati
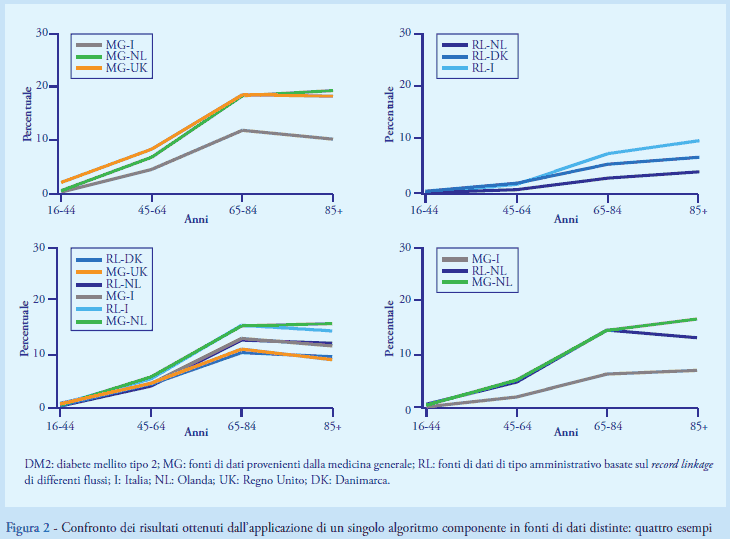
Le popolazioni di studio locali variavano da 1,4 a 3,4 milioni di soggetti nelle RL e da 1 a 3,3 milioni di soggetti circa nelle MG, per un totale di oltre 11 milioni d’individui. Il setting assistenziale in cui le diagnosi venivano registrate si associava a nette differenze dei risultati ottenuti: nella fascia d’età 45-64 la percentuale di soggetti identificati nelle MG, che disponevano di diagnosi dall’assistenza primaria, variava dal 12% in MG-NL al 18% circa in MG-UK e MG-I, mentre nelle RL, che invece disponevano di diagnosi ospedaliere, questa variava dal 3% circa in RL-NL al 7% in RL-I (Figura 2). La farmacoutilizzazione era l’unico dominio di dati disponibile in tutte le MG e RL e i componenti che utilizzavano questi dati producevano i risultati più omogenei tra tutte le 6 fonti di dati considerate. I componenti basati sull’utilizzo di test diagnostici (ad esempio, ≥2 test dell’emoglobina glicata in 1 anno per 5 anni consecutivi) estratti e testati sia in RL-I sia in RL-DK individuavano nella fonte di dati italiana delle percentuali di soggetti estremamente più elevate rispetto a quelle individuate in RL-DK, suggerendo una scarsa specificità di questi algoritmi in RL-I rispetto all’identificazione dei pazienti diabetici.
Gli algoritmi compositi scelti dagli esperti locali per l’identificazione del DM2 corrispondevano a 6 distinte combinazioni di componenti, una per ciascuna fonte di dato. L’algoritmo composito più semplice, scelto per RL-NL, utilizzava un solo componente basato sull’utilizzo di farmaci ipoglicemizzanti. In RL-I venivano raccomandati come criteri d’inclusione oltre all’algoritmo componente basato sull’uso dei farmaci ipoglicemizzanti anche i componenti basati sull’uso d’insulina, e quelli sulle diagnosi ospedaliere e sulle esenzioni. In RL-DK, si utilizzavano componenti basati sulle diagnosi ospedaliere e sull’assistenza secondaria sia per individuare i soggetti con DM2 sia per escludere quelli con diabete tipo 1 individuati attraverso l’uso di antidiabetici o sull’utilizzo di test diagnostici in determinate sequenze temporali (ad esempio, ≥2 test dell’emoglobina glicata in 1 anno per 5 anni consecutivi). In MG-UK veniva utilizzato un solo componente basato sulle diagnosi di DM2 in assistenza primaria. A quest’ultimo componente, in MG-NL si raccomandava l’aggiunta dei soggetti identificati attraverso l’uso di ipoglicemizzanti al fine di aumentare la sensibilità dell’algoritmo d’identificazione. In MG-I veniva utilizzato un componente basato sulle diagnosi di diabete tipo 1 come criterio di esclusione rispetto ai soggetti identificati attraverso le diagnosi di diabete non specificato e altri tre componenti che sfruttavano i risultati di laboratorio (ad esempio, ≥2 valori HbA1C >6,5%). In generale, i componenti che contribuivano maggiormente all’identificazione dei casi di DM2 nelle RL erano quelli basati sulle prescrizioni (81%-100% di tutti i casi), mentre nelle MG erano quelli basati sulle diagnosi (93%-100%) (Tabella 2).
Scarica la tabella (Impatto degli algoritmi componenti sulla popolazione totale di casi identificata in ciascuna fonte di dati attraverso l’applicazione dell’algoritmo composito scelto - pdf 60 kb).
Discussione e conclusioni
Attraverso la strategia degli algoritmi componenti è stato possibile identificare i casi di DM2 in 6 fonti di dati eterogenee utilizzando algoritmi d’identificazione specifici per ciascuna fonte di dati, sebbene costruiti attraverso blocchi standard. L’utilizzo di questo approccio metodologico, negli studi multidatabase, è in grado di garantire una documentazione più trasparente e accessibile degli algoritmi d’identificazione utilizzati localmente (6), oltre a permettere il confronto dei singoli algoritmi componenti attraverso fonti di dati molto diverse tra loro e fornire elementi utili per la corretta interpretazione dei risultati ottenuti. Il limite principale di questa strategia riguarda la validità degli algoritmi finali scelti. Infatti, in assenza di uno studio di validazione, la scelta dell’algoritmo deve basarsi su assunti relativi alla validità attesa dell’algoritmo stesso. Tuttavia, proprio in questi casi, la strategia qui descritta è in grado di fornire degli argomenti utili a sostenere tali assunti attraverso il confronto tra fonti di dati a livello di componenti. Nel caso del DM2, ad esempio, un algoritmo basato sull’uso di farmaci o diagnosi ospedaliere sarà certamente meno sensibile e identificherà pazienti a uno stadio più avanzato della malattia diabetica rispetto a un algoritmo basato sulle diagnosi dall’assistenza primaria dove anche i pazienti in sola dieta sono catturati. Negli studi multidatabase, questo tipo di informazioni può rivelarsi particolarmente utile per spiegare le incongruenze dei risultati ottenuti nelle Versione estesa online Inserto BEN diverse fonti di dati utilizzate (7), generando delle ipotesi che possono essere facilmente verificate attraverso analisi di sensibilità effettuate a livello di algoritmo componente.
Dichiarazione sui conflitti di interesse
Gli autori partecipano al progetto EMIF, che è finanziato dalla Innovative Medicines Initiative, un consorzio tra l’Unione Europea e la federazione europea delle aziende farmaceutiche. Inoltre, conducono studi di farmaco epidemiologia finanziati da aziende farmacitiche, aderenti al Codice di Condotta della rete europea dei centri di farmacoepidemiologia e farmacovigilanza.
Riferimenti bibliografici
1. Pathak J, Kho AN, Denny JC. Electronic health records-driven phenotyping: challenges, recent advances, and perspectives. J Am Med Inform Assoc 2013;20(e2):e206-e11.
2. Richesson RL, Horvath MM, Rusincovitch SA. Clinical research informatics and electronic health record data. Yearb Med Inform 2014;9:215-23.
3. Richesson RL, Rusincovitch SA, Wixted D, et al. A comparison of phenotype definitions for diabetes mellitus. J Am Med Inform Assoc 2013;20(e2):e319-26.
4. Morley KI, Wallace J, Denaxas SC, et al. Defining disease phenotypes using national linked electronic health records: a case study of atrial fibrillation. PLoS One 2014;9(11):e110900.
5. Gini R, Schuemie M, Brown J, et al. Data Extraction and Management in Networks of Observational Health Care Databases for Scientific Research: a Comparison of EU-ADR, OMOP, Mini- Sentinel and MATRICE Strategies. EGEMS (Wash DC) 2016;4(1):1189.
6. Benchimol EI, Smeeth L, Guttmann A, et al. The REporting of studies Conducted using Observational Routinely-collected health Data (RECORD) statement. PLoS Med 2015;12(10):e1001885.
7. Madigan D, Ryan PB, Schuemie M, et al. Evaluating the impact of database heterogeneity on observational study results. Am J Epidemiol 2013;178(4):645-51.
8. Avillach P, Coloma PM, Gini R, et al. Harmonization process for the identification of medical events in eight European healthcare databases: the experience from the EU-ADR project. J Am Med Inform Assoc 2013;20(1):184-92.
99. Roberto G, Leal I, Sattar N, et al. Identifying cases of type 2 diabetes in heterogeneous data sources: strategy from the EMIF Project. PLoS One 2016;11(8):e0160648.
10. Ryden L, Grant PJ, Anker SD, et al. ESC Guidelines on diabetes, pre-diabetes, and cardiovascular diseases developed in collaboration with the EASD: the Task Force on diabetes, pre-diabetes, and cardiovascular diseases of the European Society of Cardiology (ESC) and developed in collaboration with the European Association for the Study of Diabetes (EASD). Eur Heart J 2013;34(39):3035-87.

 Bollettino epidemiologico nazionale
Bollettino epidemiologico nazionale